Klasifikasi Aritmia Dari Hasil Elektrokardiogram ...
Transcript of Klasifikasi Aritmia Dari Hasil Elektrokardiogram ...

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer e-ISSN: 2548-964X Vol. 2, No. 3, Maret 2018, hlm. 1170-1178 http://j-ptiik.ub.ac.id
Fakultas Ilmu Komputer
Universitas Brawijaya 1170
Klasifikasi Aritmia Dari Hasil Elektrokardiogram Menggunakan Support
Vector Machine Dengan Seleksi Fitur Menggunakan Algoritma Genetika
Reiza Adi Cahya1, Candra Dewi2, Bayu Rahayudi3
Program Studi Teknik Informatika, Fakultas Ilmu Komputer, Universitas Brawijaya
Email: [email protected], [email protected], [email protected]
Abstrak
Elektrokardiogram (EKG), atau rekam jantung, dapat digunakan untuk mengenali kelainan detak
jantung atau aritmia. Bantuan komputer dengan teknik machine learning tertentu dapat digunakan untuk
mengenali aritmia secara otomatis. Tetapi data numerik yang belum diproses dari EKG mempunyai
jumlah fitur yang banyak, yang dapat mengurangi kualitas pengenalan otomatis. Algoritma genetika
(genetic algorithm, GA) dapat digunakan untuk menyeleksi fitur sehingga didapat data dengan jumlah
fitur yang lebih rendah. GA akan membuat data set dengan fitur yang sudah diseleksi, dan data set
tersebut digunakan untuk melatih support vector machine (SVM) untuk mengklasifikasikan aritmia.
Untuk pelatihan dan pengujian, digunakan data EKG dari database aritmia Massachusetts Institute of
Technology–Beth Israel Hospital (MIT-BIH). Masing-masing data merupakan rekam jantung selama 6
detik dan diklasifikasikan ke dalam detak jantung normal dan 3 jenis aritmia. Hasil yang didapat dari
penelitian menunjukkan bahwa GA-SVM mempunyai akurasi rata-rata sebesar 82.5% menggunakan
120 data latih dan 20 data uji. GA-SVM juga dapat menurunkan jumlah fitur, dari 2160 jumlah fitur
awal menjadi rata-rata 406 fitur.
Kata kunci: aritmia, elektrokardiogram, support vector machine, algoritma genetika, seleksi fitur
Abstract
Electrocardiogram (ECG) can be used to recognize abnormal heart beats or arrhythmia. Automatic
arrhythmia recognition can be achieved through the use of machine learning techniques. However, ECG
generates raw numerical data with large amount of features that can reduce the quality of automatic
recognition. Genetic algorithm (GA) can be utilized to perform a feature selection, reducing the amount
of features. Data with reduced features then will be used to train a support vector machine (SVM)
classifier. ECG data from the Massachusetts Institute of Technology–Beth Israel Hospital (MIT-BIH)
arrhythmia database is used as training and testing data. Each data is a six-second ECG recording, and
is classified into normal heartbeat and 3 different kind of arrhythmias. Result shows that GA-SVM
yielded average accuracy of 82.5% with 120 training data and 20 test data, and reduced the amount of
feature from 2160 original features to an average of 406 reduced features.
Keywords: arrhythmia, electrocardiogram, support vector machine, genetic algorithm, feature selection
1. PENDAHULUAN
Aritmia atau kelainan detak jantung telah
banyak diderita oleh penduduk dunia – salah satu
jenis aritmia, atrial fibrillation (afib) telah
menyerang 6 juta penduduk Eropa dan 2,3 juta
penduduk Amerika (Kannel & Benjamin, 2008),
sedangkan aritmia jenis ventricular tachycardia
(vtac) telah menyebabkan 300.000 kematian di
Amerika (Compton, 2015).
Aritmia menyebabkan jantung berdetak
lebih cepat, lebih lambat, atau menjadi tidak
teratur. Kondisi menyebabkan gejala-gejalan
seperti rasa lelah dan rasa sakit di dada. Untuk
mendeteksi aritmia, dokter menggunakan rekam
jantung atau elektrokardiogram (American
Health Association, 2016).
Elektrokardiogram (EKG) adalah hasil
rekaman aktivitas jantung yang didapat dengan
menempelkan elektrode ke kulit untuk
menangkap arus listrik yang dihasilkan jantung.
Deretan aktivitas-aktivitas jantung yang direkam
oleh EKG dapat digunakan sebagai indikator
adanya gangguan irama jantung, yang dapat
digunakan oleh dokter atau perawat untuk

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1171
Fakultas Ilmu Komputer, Universitas Brawijaya
mengambil tindakan yang tepat (Wilkins, 2011).
Dalam menginterpretasikan hasil EKG,
diperlukan pengetahuan yang luas karena setiap
macam aritmia memiliki ciri-ciri yang berbeda,
seperti bentuk-bentuk gelombang dan jumlah
detak jantung permenit (Wilkins, 2011).
Pengenalan manual seperti ini rentan terhadap
kesalahan diagnosis karena kurangnya ketelitian.
Praktisi-praktisi non-ahli kardiologi, seperti
perawat, juga dapat mengalami kesulitan
mengenali jenis-jenis gelombang.
Untuk membantu tenaga medis
menginterpretasikan hasil EKG, dapat
digunakan bantuan sistem komputer dengan
machine learning. Akan tetapi, kendala dalam
menerapkan pengenalan otomatis dengan sistem
komputer adalah EKG menghasilkan data
numerik dengan jumlah besar. Hal ini
dikarenakan EKG merupakan rekaman aktivitas
jantung dalam millivolt (mV) yang direkam
setiap sepersekian detik (Wilkins, 2011).
Contoh, pada database aritmia Massachusetts
Institute of Technology–Beth Israel Hospital
(MIT-BIH) rekam jantung selama 6 detik
menghasilkan titik data sebanyak 2160 buah
(Moody & Mark, 1997).
Dalam mengatasi jumlah fitur yang besar
ini, support vector machine (SVM) adalah salah
satu algoritma yang dapat digunakan. SVM
mempunyai keuntungan karena tidak begitu
dipengaruhi oleh besarnya jumlah fitur pada data
(curse of dimensionality). Selain itu SVM juga
tidak perlu mengetahui distribusi data pada
ruang fitur (Melgani & Bazi, 2008).
Untuk meningkatkan kinerja SVM, jumlah
fitur dapat dikurangi dengan melakukan proses
seleksi fitur. Pengurangan jumlah fitur dengan
memiliki keuntungan yakni mempermudah
visualisasi dan pemahaman data, mengurangi
biaya komputasi, dan meningkatkan kinerja
algoritma (Guyon & Elisseeff, 2003).
Beberapa penelitian tentang SVM dan
proses seleksi fitur telah dilakukan sebelumnya.
Melgani dan Bazi (2008) mengembangkan SVM
dengan kernel radial basis function (RBF) untuk
mengklasifikasi 5 jenis detak jantung. SVM
dioptimasi dengan particle swarm optimization
(PSO) untuk menentukan parameter C dan γ.
PSO juga digunakan untuk memilih subset fitur
yang digunakan untuk melatih SVM. Dengan
500 data latih, SVM-PSO dapat menentukan
dengan benar 90,52% data dari 40.438 total data
uji.
Dalam penelitian lain, Anbarasi, Anupriya,
dan Inyengar (2010) melakukan penelitian untuk
deteksi penyakit jantung dengan seleksi fitur
menggunakan genetic algorithm (GA).
Penelitian dilakukan dengan 3 jenis teknik
klasifikasi yakni Naïve Bayes, decision tree, dan
klasifikasi melalui clustering. Dataset terdiri dari
909 data. Setiap data memiliki 13 fitur dan 2
kelas, yakni sembuh (tidak mempunyai penyakit
jantung) dan sakit (mempunyai penyakit
jantung). GA dapat menyeleksi 6 fitur dari 13
fitur yang ada. Hasil klasifikasi dari fitur yang
sudah diseleksi sangat memuaskan, Naïve Bayes
memiliki akurasi sebesar 96,5% dan decision
tree memiliki akurasi sebesar 99,2%. Klasifikasi
melalui klustering memiliki akurasi yang cukup
bagus yakni 88,2%.
Untuk penelitian SVM tanpa seleksi fitur,
Cholissodin, dkk (2014) melakukan klasifikasi
dokumen komplain elektronik kampus dengan
directed acyclic graph (DAG) SVM dan analytic
hierarchy processing (AHP). Data-data
diklasifikaskan ke empat kelas berdasarkan
urgensi dan pentingnya komplain yang diterima.
Data-data kemudian dioleh dengan text
preprocessing. Kemudian AHP digunakan untuk
mendapat bobot setiap kelas, dan akhirnya SVM
digunakan untuk mengklasifikakan data. Hasil
yang diperoleh adalah akurasi selalu lebih tinggi
tanpa penggunaan bobot AHP dengan akurasi
terbaik senilai 82,61%.
Berdasarkan penjelasan yang telah
dipaparkan, akan dilakukan penelitian untuk
mengklasifikasikan aritmia dari hasil EKG
dengan SVM dengan proses seleksi fitur. Data
penelitian diambil dari database aritmia MIT-
BIH (Moody & Mark, 1997). Setiap data
merupakan rekam jantung selama 6 detik kanal
MLII (limb lead II yang dimodifikasi) yang
diubah menjadi 2160 fitur dan dinormalisasi
dengan metode min-max. Setiap data
mempunyai kelas yakni detak jantung normal,
atrial fibrillation, PVC bigeminy, dan
ventricular tachycardia. Dataset terdiri dari 120
data latih dan data uji. Proses seleksi fitur
dilakukan menggunakan GA. Pengujian yang
dilakukan meliputi melihat pengaruh berbagai
parameter GA dan SVM terhadap hasil
klasifikasi.
2. JANTUNG DAN ARITMIA
Jantung adalah organ penuh otot yang
terletak di dada, di belakang sternum di
mediastinum, di antara paru-paru, dan di depan
tulang belakang. Jantung terdiri dari empat
ruang, yang terdiri dari dua atria dan dua

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1172
Fakultas Ilmu Komputer, Universitas Brawijaya
ventrikel. Atria dan ventrikel masing-masing
dibagi menjadi kanan dan kiri. Atria dan
ventrikel berperan dalam siklus peredaradan
darah dalam tubuh manusia (Wilkins, 2011).
Untuk memompa darah, jantung
memerlukan impuls listrik. Aktivitas listrik
jantung ini dapat digambarkan dengan
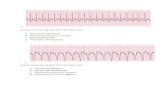
elektrokardiogam (EKG). Rekaman EKG
berbentuk gelombang yang digambarkan pada
kertas dengan kisi. Sumbu horizontal
menunjukkan waktu dan sumbu vertikal dapat
menunjukkan tegangan listrik atau amplitudo.
EKG direkam dengan menempelkan elektrode
ke kulit pasien. Tempat penempelan elektrode
mempengaruhi EKG yang dihasilkan, dan
terdapat 12 jenis rekaman EKG (Wilkins, 2011).
Dalam penelitian ini, jenis-jenis aritmia
yang akan dikenali adalah:
a. Detak jantung normal.
b. Atrial fibrillation.
c. Premature ventricular contraction
(PVC) bigeminy.
d. Ventricular Tachycardia.
3. SUPPORT VECTOR MACHINE
Misalkan terdapat sekumpulan data latih
berjumlah 𝑛 dengan fitur sejumlah 𝑚 (𝒙𝑖 ∈ℜ𝑚, 𝑖 = 1,2, … , 𝑛), support vector machine
(SVM) akan mengklasifikasikan setiap data ke
dalam target kelas yang bernilai 𝑦𝑖 ∈ (+1, −1).
Dari data latih tersebut, SVM melakukan
klasifikasi dengan menemukan bidang
(hyperplane) yang dapat memisahkan data-data
dari kedua kelas dengan margin yang paling
besar (Huang, Kecman & Kopriva, 2006).
Untuk menglasifikasikan data 𝒙 ke dalam
kelas 𝑦 ∈ (+1, −1), digunakan persamaan 1,
dengan proses penurunan persamaan 1 secara
keseluruhan dapat dilihat pada Bennett &
Campbell (2000).
𝑓(𝒙) = 𝑠𝑖𝑔𝑛(∑ 𝑦𝑖𝛼𝑖𝐾(𝒙𝒊, 𝒙) + 𝑏𝑛𝑖=1 ) (1)
Dimana 𝛼𝑖 adalah Lagrange multiplier
untuk data ke-i dan 𝑏 adalah nilai bias yang
didapat dari persamaan 2.
𝑏 = −1
2(
∑ 𝑦𝑖𝛼𝑖𝐾(𝒙𝒊, 𝒙+)𝑖∈𝑆𝑉
+ ∑ 𝑦𝑖𝛼𝑖𝐾(𝒙𝒊, 𝒙−)𝑖∈𝑆𝑉
) (2)
Dimana 𝑥+ adalah data dengan nilai 𝛼𝑖
terbesar untuk kelas +1 dan 𝑥− data dengan nilai
𝛼𝑖 terbesar untuk kelas −1. SV atau support
vectors adalah data-data yang mempunyai nilai
𝛼𝑖 lebih dari 0 (Huang, Kecman & Kopriva,
2000).
𝐾(𝒙𝒊, 𝒙𝒋) adalah fungsi kernel untuk
memetakan data untuk kasus data yang tidak
dapat dipisahkan secara linear. Fungsi kernel
yang digunakan adalah radial basis function
(RBF) pada persamaan 3.
𝐾(𝒙𝒊, 𝒙𝒋) = exp (−‖𝒙𝒊−𝒙𝒋‖
𝟐
2𝜎2 ) (3)
Nilai 𝛼𝑖 didapat dengan menyelesaikan
bidang pemisah. Pencarian bidang pemisah
tersebut merupakan masalah optimasi dan dapat
diselesaikan dengan beberapa cara. Pada
penelitian ini, cara yang digunakan adalah
sequential learning (Vijayakumar & Wu, 1999).
Sequential learning dapat menemukan bidang
pemisah optimal dengan lebih cepat dibanding
dengan metode quadraric programming yang
biasanya digunakan untuk memecahkan
optimasi.
SVM hanya dapat mengklasifikasikan data
secara biner. Untuk klasifikasi lebih dari 2 kelas,
dapat digunakan berbagai strategi memecah
klasifikasi multikelas menjadi beberapa
klasifikasi biner. Dalam penelitian ini digunakan
strategi binary decision tree (BDT) (Madzarov,
Gjorgjevikj & Chorbev, 2008). BDT mempunyai
prinsip membentuk pohon keputusan
berdasarkan jarak masing-masing kelas.
4. ALGORITMA GENETIKA
Algoritma genetika atau Genetic Algorithm
(GA) adalah algoritma optimasi (Coley, 1999)
dan pencarian stokastik (Gen & Cheng, 2000)
yang menggunakan konsep seleksi alam sebagai
dasar cara kerjanya. GA merupakan sebuah
metode umum atau framework yang dapat
digunakan untuk menyelesaikan berbagai
macam masalah (Coley, 1999).
GA bekerja dengan memanipulasi populasi
atau kumpulan individu yang merepresentasikan
solusi terhadap seuatu masalah. Pada awalnya,
populasi dibangkitkan secara acak pada berbagai
titik di ruang pencarian (Coley, 1999).
Kemudian dari populasi awal, dibentuk
individu-individu baru yang disebut dengan
offspring. Offspring dibentuk dengan proses
reproduksi yang dilakukan dengan dua cara yaitu
crossover (menggabungkan dua individu untuk
membentuk individu baru) dan mutasi
(mengubah bagian dari sebuah individu untuk
membuat individu baru). Seluruh individu
(termasuk offspring) diukur kemampuannya
dalam memecahkan masalah, yang diukur
dengan nilai fitness. Populasi baru dibentuk

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1173
Fakultas Ilmu Komputer, Universitas Brawijaya
dengan memilih individu-individu yang lebih
baik. Jika proses ini diulang selama beberapa
generasi, akan didapat individu terbaik (gBest)
yang diharapkan dapat menyelesaikan masalah
dengan baik (Gen & Cheng, 2000).
Individu memiliki deretean gen yang
disebut kromosom. Kromosom ini
merepresentasikan solusi untuk permasalahan
yang dihadapi. Proses melakukan pengubahan
dari solusi menjadi urutan gen dinamakan
encoding. Terdapat beberapa macam encoding,
yakni encoding biner, encoding bilangan real,
encoding permutasi integer, dan encoding
struktur data (Gen & Cheng, 2000).
5. SVM DENGAN SELEKSI FITUR
MENGGUNAKAN GA
GA digunakan untuk membentuk subfitur
yang optimal dari 2160 fitur awal. Untuk itu,
digunakan encoding biner. Gen ke-i setiap
kromosom merepresentasikan apakah fitur ke-i
digunakan dalam proses pelatihan SVM (nilai 1
menunjukkan fitur digunakan dan nilai 0
menunjukkan fitur tidak digunakan). Dengan
demikian, kromosom mempunyai 2160 gen.
Setiap gen dievaluasi dengan membentuk model
SVM. Nilai akurasi dan jumlah fitur digunakan
untuk menghitung nilai fitness.
Proses dari GA-SVM dapat dilihat pada
gambar 2. Penjelasan setiap langkah-langkah
GA-SVM adalah sebagai berikut:
Inisialisasi populasi: Inisialisasi populasi awal
dilakukan dengan membangkitakan nilai real
acak untuk setiap gen pada setiap kromsom.
Kemudian nilai real tersebut diubah menjadi 0
atau 1 dengan thresholding (1 jika nilai kurang
dari sama dengan threshold dan 0 jika tidak).
Nilai threshold untuk setiap kromosom dibuat
berbeda. Dengan demikian, populasi awal
memiliki jumlah fitur terseleksi yang bervariasi.
Penggunaan inisialisasi yang sederhana dengan
langsung membangkitkan nilai 0/1 membuat
semua kromosom memilih sekitar 1080 fitur
(probabilitas 0.5×2160 total fitur) sehingga
populasi awal kurang bervariasi.
Reproduksi: Reproduksi dibagi menjadi 2:
a. Crossover dilakukan dengan one-cut
point.. Crossover akan menghasilkan
subfitur yang mempunyai karakteristik
dari kedua induknya.
Gambar 1 Alur GA-SVM
b. Mutasi dilakukan dengan single
mutation. Mutasi akan menghasilkan
subfitur dengan karakteristik baru yang
tidak dimiliki induk.
𝑐𝑟 (crossover rate) dan 𝑚𝑟 (mutation rate)
adalah parameter dalam rentang [0, 1] yang
menentukan jumlah crossover dan mutasi pada 1
generasi.
Evaluasi dengan melatih SVM: Untuk
menghitung nilai fitness, dibentuk model SVM
menggunakan subfitur yang telah diseleksi.
dalam kromosom.
Model SVM dilatih dengan menggunakan
sequential learning (Vijayakumar &Wu, 1999).
Dalam penelitian ini, sequential learning
dikatakan konvergen jika iterasi maksimal telah
tercapai atau perubahan 𝛼𝑖 lebih kecil dari batas
yang telah ditentukan (max(|𝛿𝛼𝑖|) < 휀 ).
Fungsi fitness dari GA-SVM dihitung
menggunakan persamaan 7.

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1174
Fakultas Ilmu Komputer, Universitas Brawijaya
𝑓𝑖𝑡𝑛𝑒𝑠𝑠 = 0.85 × 𝑓1 + 0.15 × 𝑓2 (4)
Dimana 𝑓1 adalah akurasi dari SVM yang
telah dibentuk dengan fitur yang sudah
terseleksi. 𝑓2 adalah persentase dari fitur yang
tidak terpilih.
Seleksi: Untuk seleksi dilakukan dengan binary
tournament. 2 individu acak dipilih dari
populasi, dan individu dengan fitness terbesar
dinyatakan lolos untuk generasi selanjutnya.
Proses ini diulang sebanyak jumlah populasi.
Pembandingan solusi: Pada setiap generasi,
kromosom terbaik pada generasi ke-𝑖 (𝑔𝐵𝑒𝑠𝑡𝑖)
dibandingkan dengan kromosom terbaik pada
generasi sebelumnya (𝑔𝐵𝑒𝑠𝑡). Jika fitness
𝑔𝐵𝑒𝑠𝑡𝑖 lebih baik dari 𝑔𝐵𝑒𝑠𝑡, maka 𝑔𝐵𝑒𝑠𝑡𝑖
dinyatakan sebagai 𝑔𝐵𝑒𝑠𝑡 yang baru.
Konvergensi: GA akan berhenti dengan 2
syarat. Pertama, GA harus dijalankan minimal
25 generasi. Kedua, jika selama 10 generasi
tidak terjadi perbaikan akurasi dan fitur yang
terseleksi tidak turun lebih dari 10%. Kondisi
kedua jika dinyatakan dengan persamaan 4, akan
menghasilkan nilai threshold:
∆𝑓𝑖𝑡𝑛𝑒𝑠𝑠 = 0.85 × ∆𝑓1 + 0.15 × ∆𝑓2
= 0.85 × 0 + 0.15 × 0.1 (5)
= 0 + 0.015 = 0.015
6. HASIL DAN PEMBAHASAN
Kinerja dari GA-SVM dilihat dengan hasil
akurasi, yakni perbandingan antara data uji yang
diklasifikasikan dengan benar dan jumlah semua
data uji. Dalam penelitian ini, digunakan 120
data latih dan 20 data uji, dengan 5 data uji untuk
setiap kelas. Kemudian dilihat pengaruh
parameter-parameter GA-SVM terhadap
akurasi. Parameter-parameter yang diuji adalah:
a. Ukuran populasi GA
b. Tingkat crossover GA (𝑐𝑟)
c. Tingkat mutasi GA (𝑚𝑟)
d. Nilai threshold 휀 SVM
e. Augmenting factor 𝜆 SVM
f. Konstanta 𝐶 SVM
g. Learning rate 𝛾 SVM
h. Nilai 𝜎 dari kernel RBF
i. Jumlah iterasi SVM
Untuk setiap nilai parameter, pengujian
diulang sebanyak 10 kali untuk mendapatkan
nilai fitness rata-rata. Dalam pengujian
digunakan parameter awal sebagai berikut:
a. Ukuran populasi: 50
b. 𝑐𝑟: 0,9
c. 𝑚𝑟 0,1
d. σ dari RBF: 2
e. λ: 0,5
f. γ: 0.01
g. C: 1
h. ε: 10-5
i. Iterasi maksimal SVM: 100
6.1 Hasil dan Pembahasan Pengujian
Ukuran Populasi GA
Gambar 2 Hasil Pengujian Ukuran Populasi
Hasil dari pengujian ukuran populasi
disajikan pada gambar 3. Dari hasil pengujian
didapat bahwa nilai fitness cenderung sebanding
dengan ukuran populasi. Populasi menunjukkan
berapa banyak solusi (Gen & Cheng, 2000),
sehingga populasi yang kecil menyebabkan tidak
banyak solusi yang ditelusuri. Jumlah populasi
100 dipilih sebagai jumlah populasi yang tepat
pada masalah ini dan digunakan pada pengujian-
pengujian selanjutnya.
6.2 Hasil dan Pembahasan Pengujian
Crossover Rate dan Mutation Rate
Gambar 3 Hasil Pengujian Crossover Rate dan
Mutation Rate
Hasil dari pengujian kombinasi 𝑐𝑟 dan 𝑚𝑟
disajikan pada 4. Pengujian menunjukkan bahwa
crossover rate yang lebih besar menghasilkan
fitness yang lebih baik. Hal ini dikarenakan
masalah memiliki ruang pencarian yang besar
0,5
0,6
0,7
0,8
0,9
1
10 20 30 40 50 60 70 80 90 100
Rat
a-ra
taFitness
Ukuran Populasi
0,5
0,6
0,7
0,8
0,9
1
Rat
a-ra
taFitness
Cr/Mr

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1175
Fakultas Ilmu Komputer, Universitas Brawijaya
(22160), dan crossover memberikan kemampuan
untuk menjelahi ruang pencarian yang lebih luas
(Gen & Cheng, 2000). Nilai kombinasi
parameter 𝑐𝑟 dan 𝑚𝑟 yang dipilih untuk
pengujian selanjutnya adalah 0.9 dan 0.1.
6.3 Hasil dan Pembahasan Pengujian
Threshold 𝜺 SVM
Gambar 4 Hasil Pengujian Threshold ε SVM
Hasil dari pengujian nilai threshold ε
disajikan pada 5. Bertambahnya nilai 휀
cenderung mengakibatkan penurunan nilai
fitness. Nilai ε menunjukkan seberapa besar 𝛿𝛼𝑖
yang dibutuhkan untuk melanjutkan pencarian.
Dengan demikian, nilai 휀 yang besar akan
menyebabkan pencarian mudah terhenti karena
pembaruan 𝛼𝑖 tidak cukup besar, walaupun
konvergensi belum tercapai. Nilai 10−7
digunakan sebagai nilai ε terbaik dan digunakan
pada pengujian-pengujian selanjutnya.
6.4 Hasil dan Pembahasan Pengujian Nilai
Augmenting Factor 𝝀 SVM
Gambar 5 Hasil Pengujian Nilai λ SVM
Hasil dari pengujian nilai augmenting
factor λ dapat dilihat pada 6. Hasil yang didapat
adalah pada nilai 0.01 hingga 0.5 nilai fitness
cenderung stabil, dan nilai 𝜆 yang lebih besar
menurunkan nilai fitness. Pada sequential
learning, nilai 𝜆 yang lebih besar memberikan
bidang pemisah yang lebih mirip dengan bidang
pemisah yang didapat dengan menyelesaikan
quadratic problem, tetapi juga menyebabkan
konvergensi lebih lama (Vijayakumar & Wu,
1999), sehingga nilai 𝛼𝑖 yang didapat pada saat
iterasi terakhir tercapai tidak optimal. Oleh
karena itu, nilai 0.5 dipilih sebagai nilai 𝜆 terbaik
dan digunakan pada pengujian-pengujian
selanjutnya.
6.5 Hasil dan Pembahasan Pengujian Nilai
𝑪 SVM
Gambar 6 Hasil Pengujian Nilai C SVM
Hasil dari pengujian nilai C dapat dilihat
pada gambar 7. Hasil pengujian menunjukkan
nilai fitness mengalami peningkatan dengan
bertambahnya nilai C. Nilai C memberikan
bobot penalti yang lebih besar pada data yang
melewati bidang pemisah, sehingga SVM yang
dihasilkan dapat menghindari kesalahan
klasifikasi (Huang, Kecman & Kopriva, 2006).
Dengan demikian nilai N = 50 digunakan
sebagai nilai C terbaik dan digunakan pada
pengujian-pengujian selanjutnya.
6.6 Hasil dan Pembahasan Pengujian Nilai
Learning Rate 𝜸 SVM
Gambar 7 Hasil Pengujian Nilai γ SVM
Hasil dari pengujian nilai 𝛾 dapat dilihat
0,5
0,6
0,7
0,8
0,9
1
Rat
a-ra
taFitness
𝜀
0,5
0,6
0,7
0,8
0,9
1
0,01 0,1 0,5 1 5 10 25 50 100
Rat
a-ra
taFitness
𝜆
0,5
0,6
0,7
0,8
0,9
1
Rat
a-ra
taFitness
C
0
0,2
0,4
0,6
0,8
1
Rat
a-ra
taFitness
𝛾

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1176
Fakultas Ilmu Komputer, Universitas Brawijaya
pada gambar 8. Hasil menunjukkan nilai fitness
cenderung stabil dari 𝛾 = 10−7 hingga 𝛾 = 10−4, mencapai titik maksimal pada 𝛾 = 0.01
dan kemudian mengalami penurunan. Sequential
Learning pada dasarnya adalah gradient ascent
(Vijayakumar & Wu, 1999) sehingga learning
rate yang terlalu kecil menyebabkan optimal
lama tercapai, dan learning rate yang terlalu
besar menyebabkan 𝛼𝑖 optimal terlewati.
Berdasarkan hasil pengujian, nilai 𝛾 = 0.01
menjadi nilai yang mempunyai keseimbangan
antara kecepetan pencarian dan tidak melawati
hasil optimal. Oleh karena itu, 𝛾 = 0.01 dipilih
menjadi nilai parameter terbaik dan digunakan
pada pengujian-pengujia selanjutnya.
6.7 Hasil dan Pembahasan Pengujian Nilai
𝝈 Kernel RBF
Gambar 8 Hasil Pengujian Nilai σ Kernel RBF
Hasil dari pengujian nilai 𝜎 dapat dilihat
pada gambar 9. Nilai fitness naik dari rentang 1
hingga 2, dan setelah itu turun. Untuk 𝛾 =1/(−2𝜎), 𝛾 yang lebih besar menghasilkan
kernel yang overfit, sedangkan nilai 𝛾 yang lebih
kecil menghasilkan kernel yang underfit
(Melgani & Bazi, 2008). Karena 𝛾 berbanding
terbalik dengan 𝜎 maka nilai 𝜎 yang besar akan
menghasilkan kernel yang underfit dan nilai 𝜎
yang kecil akan menghasilkan kernel yang
overfit. Dalam pengujian ini, didapat bahwa nilai
𝛾 = 2 adalah nilai yang dapat menjaga
keseimbangan antara overfit dan underfit
sehingga mendapat fitness terbaik dan
digunakan pada pengujian selanjutnya.
6.8 Hasil dan Pembahasan Pengujian
Hasil dari pengujian jumlah iterasi SVM
dapat dilihat pada 10. Hasil pengujian
menunjukkan fitness tidak mengalami kenaikan
atau penurunan yang signifikan dengan
bertambahnya jumlah iterasi. Hal ini
menunjukkan bahwa SVM dapat mencapai
konvergensi pada 100 iterasi. Oleh karena itu,
jumlah iterasi SVM sebesar 100 dipilih sebagai
jumlah iterasi terbaik walaupun terdapat nilai
fitness yang lebih tinggi pada jumlah iterasi yang
lebih banyak. Selain itu, jumlah iterasi yang
lebih banyak menyebabkan waktu komputasi
yang lebih lama.
Jumlah Iterasi SVM
Gambar 9 Hasil Pengujian Jumlah Iterasi SVM
6.9 Validasi Pengujian
Setelah dilakukan pengujian, didapat
parameter-parameter optimal sebagai berikut:
a. Ukuran populasi: 100
b. Crossover rate: 0,9
c. Mutation rate: 0,1
d. Threshold ε: 10-7
e. Augmenting factor λ: 0,5
f. Nilai C: 50
g. Learning rate γ: 0.01
h. σ dari RBF: 2
i. Iterasi maksimal SVM: 100
Validasi pengujian dilakukan dengan
menjalankan GA-SVM dengan parameter
optimal selama 10 kali untuk melihat kestabilan
hasil GA-SVM. Hasil dapat dilihat pada tabel 1.
GA-SVM mampu menghasilkan akurasi rata-
rata sebesar 82.5.5%. GA-SVM juga dapat
menyeleksi rata-rata 406 fitur, penurunan yang
signifikan dari fitur awal yang sebanyak 2160
fitur.
Tabel 1. Hasil Akhir Pengujian
No Akurasi Jumlah Fitur
Terpilih
Fitness
1 80% 310 0.808472222
2 80% 695 0.781736111
3 80% 348 0.805833333
4 85% 396 0.845
5 90% 320 0.892777778
6 80% 306 0.80875
7 85% 393 0.845208333
8 85% 297 0.851875
9 85% 254 0.854861111
10 75% 738 0.73625
Rata-rata 82.5% 406 0.823076389
0,5
0,6
0,7
0,8
0,9
1
1 1,5 2 2,5 3 3,5 4 4,5 5
Rat
a-ra
taFitness
𝜎
0,5
0,6
0,7
0,8
0,9
1
10
0
20
0
30
0
40
0
50
0
60
0
70
0
80
0
90
0
10
00Rat
a-ra
taFitness
Jumlah Iterasi SVM

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1177
Fakultas Ilmu Komputer, Universitas Brawijaya
7. PENUTUP
Dari hasil yang didapat dalam penelitian
klasifikasi aritmia EKG dengan menggunakan
SVM dengan seleksi fitur menggunakan GA
dapata mengklasifikasikan data dengan akurasi
sebesar 82,5% dan menyeleksi rata-rata 406 dari
2160 fitur dengan 120 data latih dan 20 data uji.
Untuk terus mengembangkan penelitian ini,
terdapat beberapa hal yang dapat
dipertimbangkan. Pertama, algoritma genetika
tidak hanya sebagai metode seleksi fitur, tetapi
juga untuk optimasi parameter sehingga dapat
ditemukan parameter yang optimal secara
otomatis. Selain itu dapat ditambahkan
mekanisme random injection pada algoritma
genetika untuk mencegah kondisi local optima.
Penelitian juga dapat dikembangkan
dengan membandingkan pengaruh kernel lain
seperti kernel polinomial, dan strategi multikelas
lain seperti one-against-all dan one-against-one
terhadap hasil klasifikasi.
DAFTAR PUSTAKA
Anbarasi, M., Anupriya, E. & Iyengar, N. C. S.
N., 2010. Enhanced Prediction of Heart
Disease with Feature Subset Selection
Using Genetic Algorithm. International
Journal of Engineering Science and
Technology, 2(10), pp. 5370-5376.
American Health Association, 2016. About
Arrhythmia. [Online]
Tersedia pada: http://www.heart.org/
HEARTORG/Conditions/Arrhythmia/
AboutArrhythmia/About-Arrhythmia
_UCM_002010_Article.jsp#.WHtC
wPl97Df [Diakses 15 January 2017].
American Health Association, 2016. Why
Arrhythmia Matters. [Online]
Tersedia pada: http://www.heart.org/
HEARTORG/Conditions/Arrhythmia/
Why ArrhythmiaMatters/Why-
Arrhythmia-Matters_UCM_002023_
Article.jsp#.WHtDUfl97Dc [Diakses 15
January 2017].
Bennett, K. P. & Campbell, C., 2000. Support
Vector Machines: Hype or Hallelujah.
SIGKDD Explorations, 2(2), pp. 1-13.
Coley, D., 1999. An Introduction to Genetic
Algorithms for Scientists and Engineers.
Singapore: World Scientific Publishing
Co. Pte. Ltd.
Cholissodin, I., Kurniawati, M., Indriati &
Arwani, I., 2014. Classification of
Campus E-Complaint Documents using
Directed Acyclic Graph Multi-Class
SVM Based on Analytic Hierarchy
Process. International Conference on
Advanced Computer Science and
Information Systems (ICACSIS), 18-29
Oktober, pp. 247-253.
Compton, S. J., 2015. Ventricular Tachycardia:
Practice Essentials, Background,
Pathophysiology. [Online]
Tersedia pada: http://emedicine.
medscape.com/article/159075-overview
[Diakses 15 January 2017].
Gen, M. & Cheng, R., 2000. Genetic Algorithms
and Engineering Optimization. New
York: John Wiley & Sons.
Guyon, I. & Elisseeff, A., 2003. An Introduction
to Variable and Feature Selection.
Journal of Machine Learning Research,
Volume 3, pp. 1157-1182.
Huang, T.-M., Kecman, V. & Kopriva, I., 2006.
Kernel Based Algortihms for Mining
Huge Data Sets. Heidelberg: Springer-
Verlag Berlin Heidelberg.
Kannel, W. & Benjamin, E., 2008. Final Draft
Status of the Epidemiology of Atrial
Fibrillation. The Medical clinics of
North America, 92(1), pp. 17-ix.
Madzarov, G., Gjorgjevikj, D. & Chorbev, I.,
2009. A Multi-class SVM Classifier
Utilizing Binary Decision Tree.
Informatica, 33(2), pp. 233-241.
Melgani, F. & Bazi, Y., 2008. Classification of
Electrocardiogram Signals with Support
Vector Machines and Particle Swarm
Optimization. IEEE Transactions on
Information Technology in Biomedicine,
12(5), pp. 667-677.
Moody, G. B. & Mark, R. G., 1997. MIT-BIH
Arrhythmia Database. [Online]
Tersedia pada: https://physionet.org/
physiobank/database/mitdb/ [Diakses
16 January 2016].
Pratama, A., Cholissodin, I. & Suprapto, 2016.
Klasifikasi Kondisi Detak Jantung
Berdasarkan Hasil Pemeriksaaan
Elektrokardiografi Menggunakan
Binary Decision Tree - Support Vector
Machine (BDT-SVM). Repositori
Jurnal Mahasiswa PTIIK UB, 21(8).

Jurnal Pengembangan Teknologi Informasi dan Ilmu Komputer 1178
Fakultas Ilmu Komputer, Universitas Brawijaya
Vijayakumar, S. & Wu, S., 1999. Sequential
Support Vector Classifiers and
Regression. Proceeding International
Conference on Soft Computing (SOCO
'99), 1-4 Juni, pp. 610-619.
Wilkins, L. W., 2011. ECG Interpretation Made
Incredibly Easy. 5th ed. Pennsylvania:
Wolters Kluwer/Lippincott Williams &
Wilkins Health.